打破领域瓶颈!张锋再发Science
微生物系统是包括CRISPR在内的许多生物技术的基础,但是序列数据库的指数级增长使得很难找到以前未识别的系统。
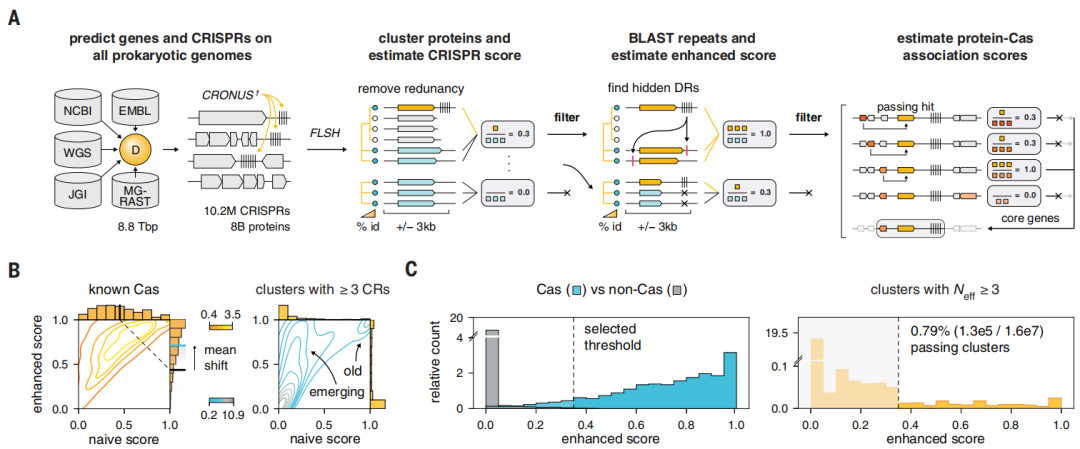
2023年11月23日,博德研究所张锋及美国国立卫生研究院Eugene V. Koonin共同通讯在Science 在线发表题为“Uncovering the functional diversity of rare CRISPR-Cas systems with deep terascale clustering”的研究论文,该研究开发了基于位置敏感哈希的快速聚类(FLSHclust)算法,该算法在线性时间内对大量数据集进行深度聚类。
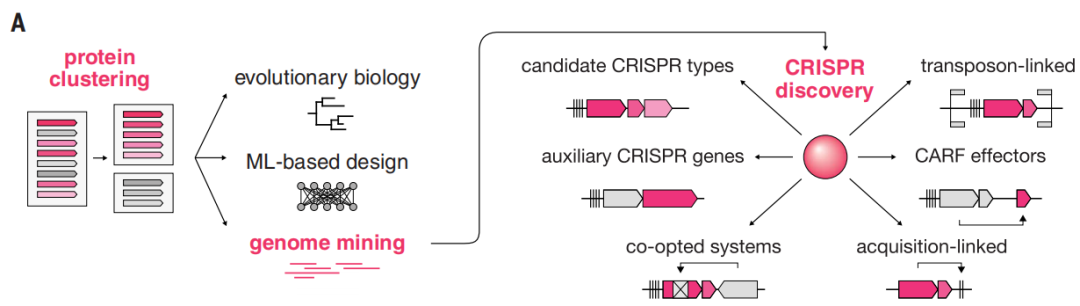
该研究将FLSHclust纳入CRISPR发现管道,并鉴定了188个以前未报道的CRISPR相关基因模块,揭示了许多与适应性免疫相关的其他生化功能。该研究通过实验表征了三种含HNH核酸酶的CRISPR系统,包括第一种具有特定干扰机制的IV型系统,并对它们进行了基因组编辑。该研究还鉴定并表征了一种候选的VII型系统,显示了它对RNA的作用。这项工作为利用CRISPR和更广泛地探索微生物蛋白质的巨大功能多样性开辟了新的途径。
最后,iNature在2023年11月18日总结了张锋在2023年发表的Science,Nature,Cell 等发表的8篇文章。
酶和天然生化系统的发现推进了分子进化研究,揭示了生物过程,为分子技术的发展提供了一个起点。在过去的几十年里,通过对快速增长的核酸和蛋白质序列数据库的系统挖掘,发现了种类繁多的蛋白质家族和功能系统。这些工作中有许多使用蛋白质聚类来对大型数据集中的相似序列进行分组。然后,这些算法的输出可用于蛋白质序列的深度学习、三维(3D)蛋白质结构预测和基因组挖掘。后者的一个主要例子是发现了以前未知的CRISPR系统,它导致了变革性生物技术和治疗方法的发展。

赞一个

IEEE Xplore,EI Compendex,Scopus
IEEE出版|2026年区块链技术与基础模型国际学术会议(BTFM 2026)IEEE出版
前沿会议

EI Compendex,Scopus
IOP-JPCS出版|2026年高端装备与智能机器人国际学术会议 (ICAEIR 2026)交叉学科
官方推荐

EI Compendex,Scopus,IEEE Xplore
IEEE出版 | 2026年计算智能与机器学习国际学术会议(CIML 2026)早鸟价
官方推荐




